Bioinformatika

Bioinformatika (basa Inggris: bioinformatics) iku (èlmu kang nyinaoni) panrapan tèhnik komputasional kanggo nglola lan nganalisis informasi biologis. Babagan iki nyakup panrapan métodhe-métodhe matématika, statistika, lan informatika kanggo mecahaké masalah-masalah biologis, mligi kanthi migunakaké sekuens DNA lan asem amino sarta informasi kang ana kaitané. Conto topik utama babagan iki ngambah basis data kanggo ngemunah informasi biologis, panyejajaran sekuens (sequence alignment), prédiksi struktur kanggo ngramalaké wangun struktur protéin uga struktur sékundhèr RNA, analisis filogenetik, lan analisis èksprèsi gen.
Sajarah
[besut | besut sumber]Istilah bioinformatics wiwit diwedharaké ing tengah éra 1980-an kanggo ngacu panrapan komputer ing biologi. Éwadéné mangkono, panrapan babagan-babagan ing bioinformatika (kaya panggawéan basis data lan pangembangan algoritma kanggo analisis sekuens biologis) wis dilakokaké wiwit taun 1960-an.
Kamajuan tèhnik biologi molekular kanggo ndungkap sekuens biologis saka protéin (wiwit awal 1950-an) lan asem nukleat (wiwit 1960-an) miwiti mekaring basis data lan tèhnik analisis sekuens biologis. Basis data sekuens protéin wiwit dikembangaké taun 1960-an ing Amérikah Sarékat, sauntara basis data sekuens DNA dikembangaké akir 1970-an ing Amérikah Sarékat lan Jerman (ing European Molecular Biology Laboratory, Laboratorium Biologi Molekular Éropa). Panemon tèhnik sekuensing DNA kang luwih rikat ing tengah taun 1970-an dadi landhesan dumadiné ledakan cacahé sekuens DNA kang kasil didungkapaké ing taun 1980-an lan 1990-an, dadi salah sawijining pambuka dalan kanggo proyèk-proyèk pandungkapan genom, ngundhakaké kabutuhan marang panglolaan lan analisis sekuens, lan pungkasané njalari lairé bioinformatika.
Mekaré internèt uga njurung mekaré bioinformatika. Basis data bioinformatika kang gumandhéng liwat internèt nggampangaké èlmuwan nglumpukaké asil sekuensing nuju basis data mau uga olèh sekuens biologis minangka bahan analisis. Saliyané iku, panyebaran program-program aplikasi bioinformatika liwat internèt nggampangaké èlmuwan ngakses program-program mau lan banjur nggampangaké pangembangané.
Panrapan utama bioinformatika
[besut | besut sumber]Basis data sekuens biologis
[besut | besut sumber]Selaras kaliyan jinis informasi biologis kang disimpen, basis data sekuens biologis bisa wujud basis data primer kanggo nyimpen sekuens primer asem nukleat utawa protéin, basis data sékundhèr kanggo nyimpen motif sekuens protéin, lan basis data struktur kanggo nyimpen data struktur protéin utawa asem nukleat.
Basis data utama kanggo sekuens asem nukleat saiki yaiku GenBank (Amérikah Sarékat), EMBL (Éropa), lan DDBJ (DNA Data Bank of Japan, Jepang). Katelu basis data mau makarya bebarengan lan ijol-ijolan data saben dina kanggo njaga kawiyaran cakupan saben basis data. Sumber utama data sekuens asem nukleat yaiku submisi langsung saka periset individual, proyèk sekuensing genom, lan pandaftaran paten. Isi saliyané sekuens asem nukleat, entri ing basis data sekuens asem nukleat lumrahé ngandhut informasi ngenani jinis asem nukleat (DNA utawa RNA), jeneng organisme sumber asem nukleat mau, lan pustaka kang ana kaitané karo sekuens asem nukleat mau.
Sauntara iku, conto sapérangan basis data wigati kang nyimpen sekuens primer protéin yaiku PIR Archived 2006-01-11 at the Wayback Machine. (Protein Information Resource, Amérikah Sarékat), Swiss-Prot (Éropa), lan TrEMBL (Éropa). Katelu basis data mau wis digabungaké ing UniProt (kang dana mligi saka Amérikah Sarékat). Entri ing UniProt ngandhut informasi ngenani sekuens protéin, jeneng organisme sumber protéin, pustaka kang ana kaitané, lan komentar kang lumrahé isiné panjlasan ngenani fungsi protéin mau.
BLAST (Basic Local Alignment Search Tool) wujud perkakas bioinformatika kang gumandhéng raket karo panggunaan basis data sekuens biologis. Panlusuran BLAST (BLAST search) ing basis data sekuens mungelaké èlmuwan kanggo nggolèki sekuens asem nukleat uga protéin kang mèmper karo sekuens tinentu kang diduwèni. Bab iki migunani umpamané kanggo nemu gen sajenis ing sapérangan organisme utawa kanggo mriksa kaabsahan asil sekuensing uga kanggo mriksa fungsi gen asil sekuensing. Algoritma kang ndhasari makarya BLAST yaiku panyejajaran sekuens.
PDB Archived 2008-08-28 at the Wayback Machine. (Protein Data Bank, Bank Data Protein) yaiku basis data tunggal kang nyimpen modhèl struktural telung dhimènsi protéin lan asem nukleat asil panemton eksperimental (kanthi kristalografi sinar-X, spektroskopi NMR lan mikroskopi èlèktron). PDB nyimpen data struktur minangka koordinat telung dhimènsi kang nggambaraké posisi atom-atom ing njero protéin utawa asem nukleat.
Panyejajaran sekuens
[besut | besut sumber]Panyejajaran sekuens (sequence alignment) yaiku prosès panyusunan/pangaturan loro utawa luwih sekuens saéngga pepadhan sekuens-sekuens mau katon nyata. Asil saka prosès mau uga ingaran minangka sequence alignment utawa alignment waé. Larik sekuens ing sawijining alignment diwènèhi sisipan (lumrahé kanthi tandha "–") saéngga kolom-kolomé ngamot karakter kang identik utawa padha ing antarané sekuens-sekuens mau. Ing ngisor iki conto alignment DNA saka rong sekuens cendhak DNA kang béda, "ccatcaac" lan "caatgggcaac" (tandha "|" nedahaké kacocokan utawa match ing antarané loro sekuens iku).
ccat---caac | || |||| caatgggcaac
Sequence alignment wujud métodhe dhasar ing analisis sekuens. Métodhe iki minangka nyinaoni évolusi sekuens-sekuens saka leluhur kang padha (common ancestor). Ora cocoké (mismatch) ing alignment diasosiasikaké kanthi prosès mutasi, éwadéné kasenjangan (gap, tandha "–") diasosiasikaké kanthi prosès insersi utawa delesi. Sequence alignment mènèhi hipotèsis marang prosès évolusi kang dumadi ing sekuens-sekuens mau. Umpamané, loro sekuens ing conto alignment ing ndhuwur bisa dadi évolusi saka sekuens kang padha "ccatgggcaac". Ing kaitané karo bab iki, alignment uga bisa nedahaké posisi-posisi kang dipertahankan (conserved) suwéné évolusi ing sekuens-sekuens protéin, kang nedahaké yèn posisi-posisi mau bisa dadi wigati kanggo struktur utawa fungsi protéin mau.
Saliyané iku, sequence alignment uga dipigunaaké kanggo nggolèki sekuens kang mèmper utawa padha ing basis data sekuens. BLAST yaiku métodhe alignment kang asring dipigunaaké ing panlusuran basis data sekuens. BLAST migunakaké algoritma heuristik ing panyusunan alignment.
Sapérangan métodhe alignment liya kang ndhisiki BLAST yaiku métodhe "Needleman-Wunsch" lan "Smith-Waterman". Métodhe Needleman-Wunsch minangka nyusun alignment global ing antarané loro utawa luwih sekuens, yaiku alignment kanggo sadawané sekuens mau. Métodhe Smith-Waterman ngasilaké alignment lokal, yaiku alignment kanggo pérangan-pérangan njero sekuens. Kaloro métodhe mau nerapaké pamrograman dinamik (dynamic programming) lan mung efektif kanggo alignment rong sekuens (pairwise alignment)
Clustal yaiku program bioinformatika kanggo alignment multipel (multiple alignment), yaiku alignment sapérangan sekuens sisan. Loro varian utama Clustal yaiku ClustalW lan ClustalX Archived 2008-02-25 at the Wayback Machine..
Métodhe liya kang bisa ditrepaké kanggo alignment sekuens yaiku métodhe kang gegandhéngan karo Hidden Markov Modhèl ("Modhél Markov Kadhelikaké", HMM). HMM iku modhèl statistika kang mula bukané dipigunaaké ing èlmu komputer kanggo ngenali wicaraning Manungsa (speech recognition). Saliyané dipigunaaké kanggo alignment, HMM uga dipigunaaké ing métodhe-métodhe analisis sekuens liyané, kaya prediksi laladan pawènèh kodhe protéin ing genom lan prédhiksi struktur sékundhèr protéin.
Prédhiksi struktur protéin
[besut | besut sumber]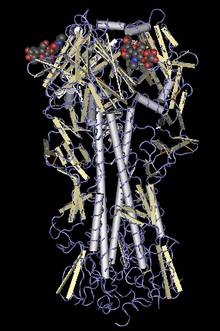
Sacara kimia/fisika, wangun struktur protéin didungkap kanthi kristalografi sinar-X utawa kanthi spektroskopi NMR, nanging rong métodhe iki banget ngentèkaké wektu lan rélatif larang. Sauntara iku, métodhe sekuensing protéin rélatif luwih gampang ndungkap sekuens asem amino protéin. Prédhiksi struktur protéin ngupaya ngramalaké struktur telung dhimènsi protéin dhedhasar sekuens asem aminoné (kanthi tembung liya, ngramalaké struktur tersier lan struktur sékundhèr dhedhasar struktur primer protéin). Lumrahé, métodhe prédhiksi struktur protéin kang ana saiki bisa dikategoriaké dadi rong golongan, yaiku métodhe pamodhèlan protéin komparatif lan métodhe pamodhèlan de novo.
Pamodhèlan protéin komparatif (comparative protéin modelling) ngramalaké struktur sawijining protéin dhedhasar struktur protéin liya kang wis dingertèni. Salah siji panrapan métodhe iki yaiku pamodhèlan homologi (homology modelling), yaiku prédhiksi struktur tersier protéin dhedhasar pepadhan struktur primer protéin. Pamodhèlan homologi didhasaraké ing téyori yèn rong protéin kang homolog duwé struktur kang mèmper banget siji lan sijiné. Ing métodhe iki, struktur sawijining protéin (ingaran protéin target) ditemtokaké dhedhasar struktur protéin liya (protéin templat) kang wis dingertèni lan duwé kamèmperan sekuens karo protéin target mau. Saliyané iku, panrapan liya pamodhèlan komparatif yaiku protéin threading kang didhasaraké ing kamèmperan struktur tanpa kamèmperan sekuens primèr. Latar wingking protéin threading yaiku struktur protéin luwih dikonservasi tinimbang sekuens protéin suwéné évolusi; laladan-laladan kang wigati kanggo fungsi protéin dipertahankan strukturé. Ing pandekatan iki, struktur kang paling kompatibel kanggo sawijining sekuens asem amino dipilih saka kabèh jinis struktur telung dhimènsi protéin kang ana. Métodhe-métodhe kang kagolong ing protéin threading ngupaya nemtokaké tingkat kompatibilitas mau.
Ing pandekatan de novo utawa ab initio, struktur protéin ditemtokaké saka sekuens primèré tanpa mbandhingaké karo struktur protéin liya. Ana akèh kamungelan ing pandekatan iki, umpamané kanthi nirokaké prosès panglipetan (folding) protéin saka sekuens primèré dadi struktur tersieré (umpamané kanthi simulasi dinamika molekular), utawa kanthi optimisasi global fungsi ènergi protéin. Prosedur-prosedur iki luwih mbutuhaké prosès komputasi kang intens, saéngga saiki mung dipigunaaké nalika nemtokaké struktur protéin-protéin cilik. Sapérangan usaha wis dilakokaké kanggo ngatasi kakurangan sumber daya komputasi mau, umpamané kanthi superkomputer (umpamané superkomputer Blue Gene [1] saka IBM) utawa komputasi kadistribusi (distributed computing, umpamané proyèk Folding@home) uga komputasi grid.
Analisis èksprèsi gen
[besut | besut sumber]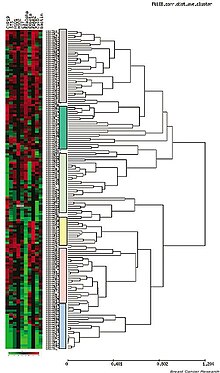
Ekspresi gen bisa ditemtokaké kanthi ngukur kadar mRNA kanthi manéka warna tèhnik (umpamané kanthi microarray utawa uga Serial Analysis of Gene Expression ["Analisis Serial Ekspresi Gen", SAGE]). Teknik-tèhnik mau lumrahé diterapaké ing analisis èksprèsi gen skala gedhé kang ngukur èksprèsi akèh gen (malah uga genom) lan ngasilaké data skala gedhé. Métodhe-métodhe panggolèkan data (data mining) ditrepaké ing data mau supaya olèh pola-pola informatif. Minangka conto, métodhe-métodhe komparasi dipigunaaké kanggo mbandhingaké èksprèsi ing antarané gen-gen, sauntara métodhe-métodhe klastering (clustering) dipigunaaké kanggo martisi data mau andhedhasar pepadhan èksprèsi gen.
Bioinformatika ing Indonésia
[besut | besut sumber]Saiki mata ajaran bioinformatika uga mata ajaran kanthi momotan bioinformatika wis diwulangaké ing sapérangan pawiyatan luhur ing Indonésia. Sekolah Èlmu lan Teknologi Hayati ITB nawakaké mata kuliah "Pengantar Bioinformatika" kanggo program Sarjana lan mata kuliah "Bioinformatika" kanggo program Pascasarjana. Fakultas Teknobiologi Universitas Atma Jaya, Jakarta nawakaké mata kuliah "Pengantar Bioinformatika". Mata kuliah "Bioinformatika" diwulangaké ing Program Pascasarjana Kimia Fakultas MIPA Universitas Indonésia (UI), Jakarta. Mata kuliah "Proteomik lan Bioinformatika" kalebu ing kurikulum program S3 bioteknologi Universitas Gadjah Mada (UGM), Yogyakarta. Materi bioinformatika kalebu ing njeron silabus sapérangan mata kuliah kanggo program sarjana uga pascasarjana biokimia,biologi, lan bioteknologi ing Institut Pertanian Bogor (IPB). Saliyané iku, riset-riset kang ngarah ing bioinformatika uga wis ditindakaké déning mahasiswa program S1 Èlmu Komputer uga program pascasarjana biologi sarta bioteknologi IPB.
Riset bioinformatika protéin ditindakaké minangka péranganing aktivitas riset rékayasa protéin ing Laboratorium Rekayasa Protein, Pusat Panlitèn Bioteknologi Lembaga Èlmu Pangetauan Indonésia (LIPI), Cibinong, Bogor. Lembaga Biologi Molekul Eijkman, Jakarta, kanthi mirunggan nduwé laboratorium bioinformatika minangka fasilitas panunjang kagiyatan riseté. Saliyané iku, basis data sekuens DNA mikroorganisme asli Indonésia gèk dikembangaké ing UI.
Pirsanana uga
[besut | besut sumber]Réferénsi lan wacan terusan
[besut | besut sumber]- Attwood, T.K., dan D.J. Parry-Smith. 1999. Introduction to Bioinformatics. Harlow: Pearson Education. ISBN 0-582-32788-1
- Krane, D.E., dan M.L. Raymer. 2003. Fundamental Concepts of Bioinformatics. San Francisco: Benjamin Cummings. ISBN 0-8053-4633-3
- Mount, D.W. 2001. Bioinformatics: Sequence and Genome Analysis. Cold Spring Harbor: Cold Spring Harbor Laboratory Press. ISBN 0-87969-608-7
Pranala njaba
[besut | besut sumber]- Daftar pitakonan sing kerep muncul ngenani bioinformatika
- Bioinformatika lan bioteknologi Archived 2007-01-03 at the Wayback Machine. (déning Arief B. Witarto, panliti LIPI)
- Jurnal Bioinformatics, salah siji jurnal èlmiah kang focus ing tema bioinformatika
- International Society for Computational Biology (ISCB)
- Asia Pacific Bioinformatics Network (APBioNet)
- Tutorial bioinformatika kanggo pamula (migunaaké alat-alat bioinformatika sing sumadiya ing internèt) Archived 2008-04-17 at the Wayback Machine.
- Download matèri bioinformatika (S-Star Bioinformatics Education)
- Planet.Bioinformatics | Agregasi blog bioinformatika[pranala mati permanèn]
